Métodos para estudiar interacciones entre proteínas y su posible aplicación al estudio de las moléculas reguladas por CCR7 en las células dendríticas
Las proteínas son las moléculas encargadas de ejecutar las funciones celulares, y, para realizar esto de forma coordinada, deben interactuar entre sí estableciendo interacciones proteína-proteína (IPPs) para constituir complejos moleculares con nuevas propiedades funcionales. De acuerdo con esto, es coherente que entorno al 80% de las proteínas interactúen entre sí para desempeñar funciones como la regulación génica o la señalización celular. Además, estas interacciones son de carácter dinámico y responden a estímulos tanto intracelulares como extracelulares.
Las interacciones proteína-proteína (IPPs) constituyen una de las 3 redes que conforman el interactoma, conjunto de interacciones entre componentes celulares, junto a las redes metabólicas que regulan todas las reacciones químicas y metabólicas que ocurren en la célula, y las redes de regulación génica, implicadas en el control de expresión génica.
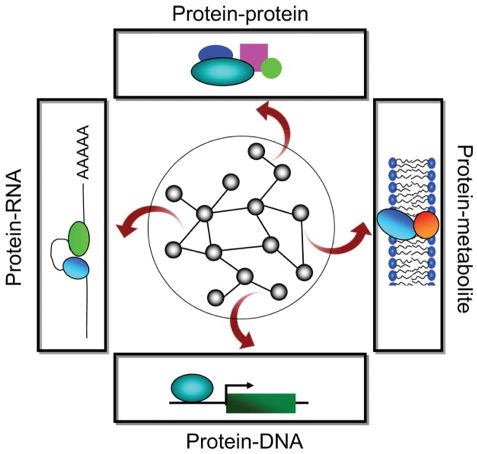
El gran reto de las proteínas
Conocer con gran precisión este interactoma es uno de los retos más importantes en biología desde la secuenciación del genoma humano en el 2003. La importancia de adquirir un mapa completo y preciso del interactoma reside en que nos permitiría comprender con mayor detalle las bases moleculares de la enfermedad humana (ya que se ha observado que en la mayoría de las enfermedades humanas el interactoma parece estar alterado) y así poder desarrollar nuevas estrategias terapéuticas dirigidas a las interacciones que se vean afectadas, consiguiendo así una medicina personalizada.
Para la identificación y caracterización de estas IPPs se han desarrollado una amplia variedad de tecnologías en los últimos 20 años, cada una de ellas con sus ventajas y limitaciones, de modo que debemos conocer con gran detalle qué tipo de interacción queremos detectar, así como su naturaleza y el tipo celular. Los métodos para detectar interacciones proteína-proteína podemos dividirlos en métodos binarios y métodos co-complejos. Los primeros tienen como objetivo detectar parejas de proteínas que interactúan mientras que los métodos co-complejos grandes complejos proteicos.
Función intracelular
Estas técnicas pueden emplearse para detectar todo tipo de interacciones entre proteínas, pero son especialmente útiles en las vías de señalización intracelular, donde el número de proteínas que interactúan es enorme. Un ejemplo de aplicación es la proteína CCR7, un receptor de quimioquinas presente en células dendríticas que regula diversas funciones agrupadas en 4 módulos de señalización (supervivencia, apoptosis, quimiotaxis y dinámica de actina) con un alto grado de independencia entre ellos.
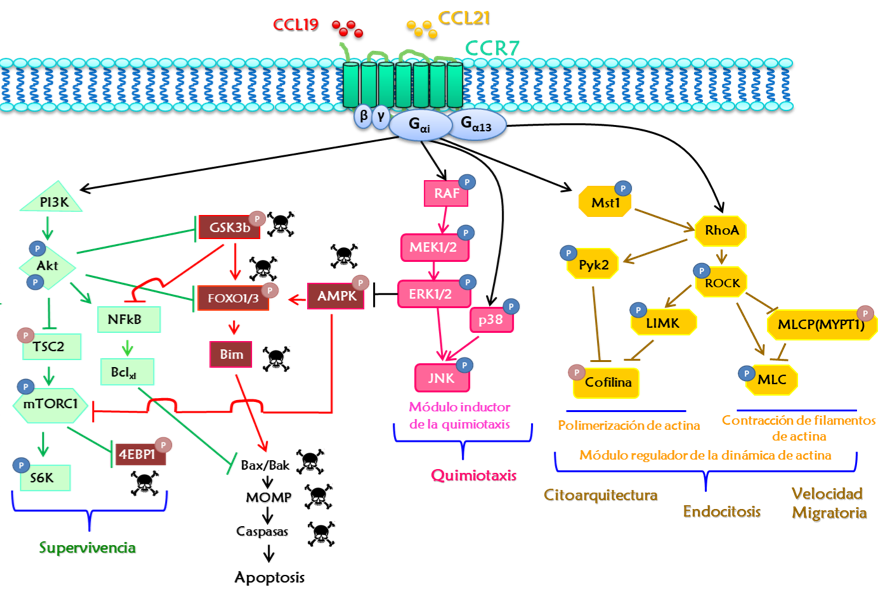
Mediante técnicas experimentales empleando inhibidores, en los últimos 10 años en el grupo de Jose Luis Rodríguez Fernández se han ido identificando las proteínas que conforman estos módulos, así como las interacciones entre ellas. Empleando el programa informático STRING se realizó una predicción de las interacciones de cada una de las proteínas pertenecientes a los módulos regulados por CCR7, obteniendo así un interactoma de cada una de ellas.
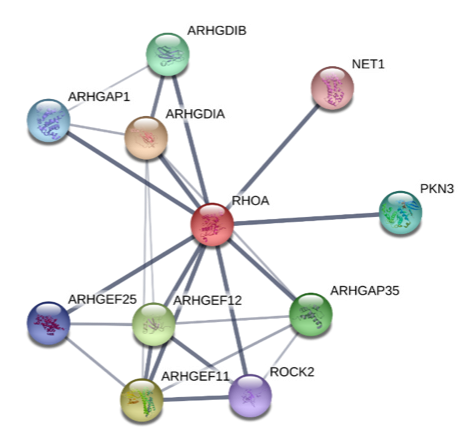
Además de analizar el interactoma individual de cada proteína, se llevó a cabo un análisis del interactoma global de CCR7, incluyendo así tanto interacciones que se producen dentro de un módulo como aquellas que se establecen entre módulos diferentes. Todo este estudio con STRING nos ha permitido confirmar las interacciones que ya habían sido detectadas experimentalmente, así como descubrir nuevas interacciones que pueden ser analizadas en futuros estudios.
Autor: Álvaro Gómez Morón.


